第四回 被骨组织包围的内耳成像用透明化方法
——Modified ScaleS
![]()
东京大学研究生院医学系研究科 浦田 真次,冈部 繁男
◆研究背景
本项研究的研究目的是开发观察耳蜗所有毛细胞的方法。耳蜗由功能不同的细胞复杂排列组成,是构造极为精密的组织,且因其被骨囊环绕,所以分析方法有限。过去分析耳蜗毛细胞组织主要通过制备切片或耳蜗铺片进行观察1,2) 。然而,由于制备切片是在耳蜗轴与水平面上进行的,虽然可以观察柯蒂氏器到侧壁的范围,但毛细胞的可观察范围十分受限。
另一方面,进行年幼小鼠的耳蜗铺片时,虽然可以观察到大部分的毛细胞,但是不可以观察被剥离的耳蜗侧壁1) 。在进行成年小鼠的耳蜗铺片时,伴随成长不仅需要通过包含蜗轴周边的骨头在内的骨囊骨化来分割耳蜗,在去除碎片化的耳蜗侧壁时还会牺牲部分毛细胞。此外,前庭膜和盖膜的去除处理比侧壁处理更需要极其精细的技术,因此该方法仅适用于熟练的研究人员。
为阐明耳蜗正常生理及病理生理结构,在保留耳蜗3D结构的状态下进行分析是必须的,完善“耳蜗解剖学限制”和“研究人员的技术限制”的技术开发势在必行。
◆试图透明化内耳的背景
在内耳耳蜗内有一种名为柯蒂氏器的感觉器官,而评价该组织重要的细胞因子,即毛细胞的感觉细胞功能对评价人耳功能极为重要。在柯蒂氏器中,响应不同频率声音的毛细胞从高音到低音有序地排列,若我们可以制备大部分细胞整体的空间图谱, 便可以将其广泛应用于失聪等耳部疾病的研究中。
然而,迄今调查柯蒂氏器的毛细胞状态的方法主要是使用组织切片分析细胞整体的一小部分。虽然还有通过人手将细小的柯蒂氏器从复杂的骨迷路中取出的方法,但由于该方法难以将精细的柯蒂氏器完好无损地取出,因此并不能用作普通的分析方法。
在此背景下,为推动耳部功能研究的发展,在无损的状态下成像完整的柯蒂氏器并检测出内在所有的毛细胞,再根据各个细胞水平判断该细胞是否受损的技术十分必要。
◆从透明化所有内耳的感觉细胞中所获得的新发现
本项研究提出了一种可以高精度地自动检测柯蒂氏器中存在的数千个毛细胞,并评价每个细胞受损程度的技术3) 。为实现这种分析,我们改良了组织透明化方法新技术,使其可应用于被骨围绕的柯蒂氏器。结果显示,通过搭配可检测组织深处荧光信号的双光子显微镜成像,成功获得了包含柯蒂氏器在内的整个内耳组织的3D图像。
接下来,我们运用机器学习方法,开发了一个可以从获得的3D图像中自动提取所有细胞位置与每个细胞信息的程序。至今为止都是使用人眼对每个细胞进行鉴定以及测量,现在通过这个程序实现了自动化,从制备样品到获得全部细胞数据仅需5天即可完成。该自动化程序可以代替人眼检测毛细胞以及识别由于细胞死亡而导致细胞脱落的位置,其准确度和精度与人工检测的水平相当。
采用新型柯蒂氏器细胞图谱制备法,可以在短时间内详细分析由于老龄或噪音引起的柯蒂氏器损伤,我们还发现了根据不同的受损原因,引起细胞死亡的细胞的空间位置分布也有所不同。另外,该方法不仅可以识别细胞,还能检测细胞内的结构以及特定的蛋白、毛细胞和神经细胞形成的突触结构。作为分析的应用实例之一,我们发现受损死亡的细胞周围的细胞也在细胞骨架水平上发生了变化。
◆透明化操作方案与应用数据
Modified ScaleS操作方案4)
A. 切除耳蜗
a. 使用4%多聚甲醛(PFA)进行心内灌注后,切除颞骨。将暴露在颅骨底部的半规管作为标记来识别内耳(图1A)。
a. 沿着骨裂切除内耳(图1B),然后将切除的内耳浸在4%PFA中并在4°C下孵育过夜。
b. 使用磷酸盐缓冲液(PBS)清洗3次,每次15 min。
c. 使用乙二胺四乙酸(EDTA)在37°C下孵育45 h。
d. 使用磷酸盐缓冲液(PBS)清洗3次,每次15 min。
e. 去除粘附在内耳周围的组织。
f. 分离半规管及前庭,切除耳蜗(图1C、D)
B. 脱脂
a. 浸于Solution 1并在37°C下孵育2h。
C. 免疫组织染色(GFP表达样品跳过该步骤)
a. 替换为含有0.1% Triton X-100的PBS(PBST)并清洗30 min。
b. 一抗在37°C下孵育2~48 h。
c. 使用PBST清洗3次,每次15 min。
d. 二抗在37°C下孵育12~48 h后,使用PBST清洗3次,每次15 min。
D. 准备样品
a. 制作圆柱状的Blu-Tack(图2(i))。
a. 注1) 圆直径的长度应与耳蜗放在载玻片上的高度相同(或圆柱略高)。
b. 在载玻片上使用Basukoku呈马蹄形涂抹(图2(ii))。
c. 将D-a中制备的Blu-Tack放在马蹄形的Basukoku上(图2(iii、iv))。
d. 在马蹄形的Blu-Tack内侧的载玻片上涂抹少量Basukoku(图2(v))。
e. 将耳蜗放在已涂抹的Basukoku上(图2(vi))。
a. 注2) 为观察所有毛细胞,请尽可能将耳蜗轴垂直于载玻片(图3)。
f. 在Blu-Tack上放置盖玻片(图2(vii)),并压迫盖玻片使其与耳蜗表面接触(图2(vii-a))。
a. 注3) 可通过目测确认盖玻片与耳蜗表面接触。为防止盖玻片破损,请小心均匀地施加压力。若盖玻片破损需要使用新的载玻片从D-a步骤重新开始。
a. 注4) 样品固定不充分时可能会无法成像,因此D-f步骤至关重要。
g. 沿载玻片填充Solution 2(图2(viii))。
a. 注5) 样品松动时请吸走Solution 2并从D-f步骤重新开始。
h. 用Basukoku密封(图2(ix))。
a. 注6) 气泡不仅会影响成像视野,也会导致样品在保存时变干燥,因此请注意不要混入气泡。
a. 注7) Solution 2浸透后,快速开始透明化。约20 min完成透明化。在自然光下,会残留耳蜗轮廓和血管纹的颜色。将手指置于载玻片后能透视的话就可以进行成像。
制备试剂
A. Solution 1(20 mL)
5.7 g盐酸胍
7 g D-山梨糖醇
3 g D-葡萄糖
800 μL(w/v)含Triton X-100的PBS(pH 6.0-8.0)
B. Solution 2(20 mL)
5.7 g盐酸胍(或4.8 g尿素)
12 g D-山梨糖醇
20 μL含Triton X-100的PBS(pH 7.1)
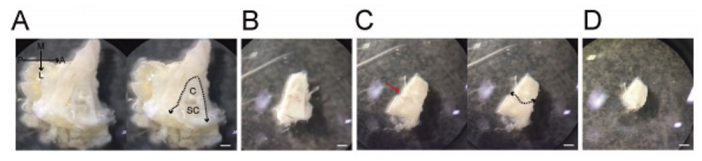
图1. 切除耳蜗
A:切除颞骨后观察颅底时可观察到球状的半规管(SC)。耳蜗(C)在SC内侧。沿内耳外围(点线)剥离组织切除内耳。B:切除内耳图。C:沿耳蜗与半规管间的缝隙(红色箭头)切割为两半,切除耳蜗。D:切除耳蜗图。比例尺 : 1 mm使用显微镜:Nikon A1MP使用镜头:N25X-APO-MP(NA1.10, WD2.00), VC 20x(NA0.75, WD1.00)
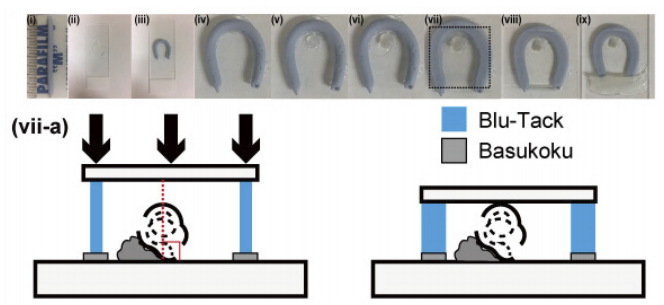
图2. 准备样品的步骤
在涂抹Basukoku的载玻片上使用Blu-Tack配合样品高度制作纵梁,并将样品放置在Basukoku上(尽量让耳蜗轴垂直于载玻片(vii-a))。然后将盖玻片放在耳蜗表面并压迫,使用Solution 2填充密封。
使用显微镜:Nikon A1MP使用镜头:N25X-APO-MP(NA1.10, WD2.00), VC20x(NA0.75, WD1.00)
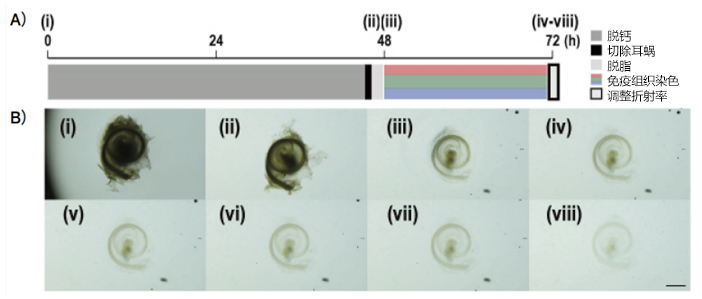
图3. Modified ScaleS
时间变化(A)、透明度变化(B)。(iv)至(viii)显示Solution 2渗透后每5min的变化。
比例尺: 1mm使用显微镜:Nikon A1MP使用镜头:N25X-APO-MP(NA1.10, WD2.00), VC 20x(NA0.75, WD1.00)
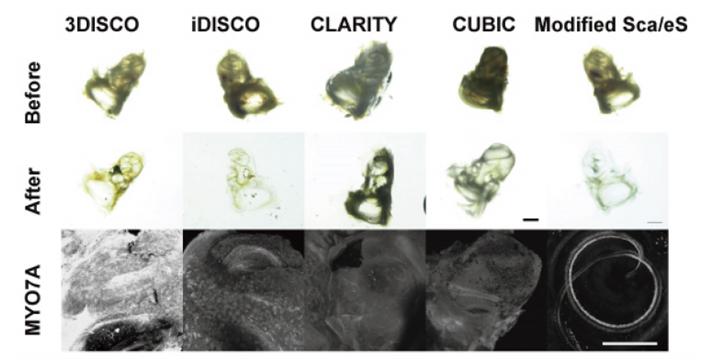
图4. 与传统方法比较
仅在Modified ScaleS中可以观察到毛细胞(MYO7A)。
比例尺: 500 μm使用显微镜:Nikon A1M使用镜头:N25X-APO-MP(NA1.10, WD2.00), VC 20x(NA0.75, WD1.00)
◆各种透明化技术的比较案例(图4)
在传统的透明化方法(3DISCO5)、 iDISCO6)、 CLARITY7)、CUBIC8))中虽然耳蜗透明度上升,但无法观察到MYO7A标记的耳蜗毛细胞。使用Modified ScaleS则可以观察到呈螺旋状排列的毛细胞群。
◆应用透明化技术的未来展望
该研究成果使我们可以在短时间内全面且单个细胞水平分析迄今为止难以分析的失聪模型动物的内耳中所发生的变化。尽管有许多以失聪为表型的疾病动物模型,但由于至今一直没有能够高效检测柯蒂氏器中毛细胞病理的技术,因此仍未能阐明细胞水平初期会发生什么变化。通过活用本次研究成果,我们便可以查明各种失聪模型动物中毛细胞的初期变化。
现在,我们发现使用Modified ScaleS观察毛细胞以外的耳蜗内细胞群也同样有效(图5)。通过研究各种失聪模型小鼠,有望在将来能够加速阐明幼儿的先天性失聪以及老年人的老年性耳聋的病理,并以此为基础开发治疗方案。
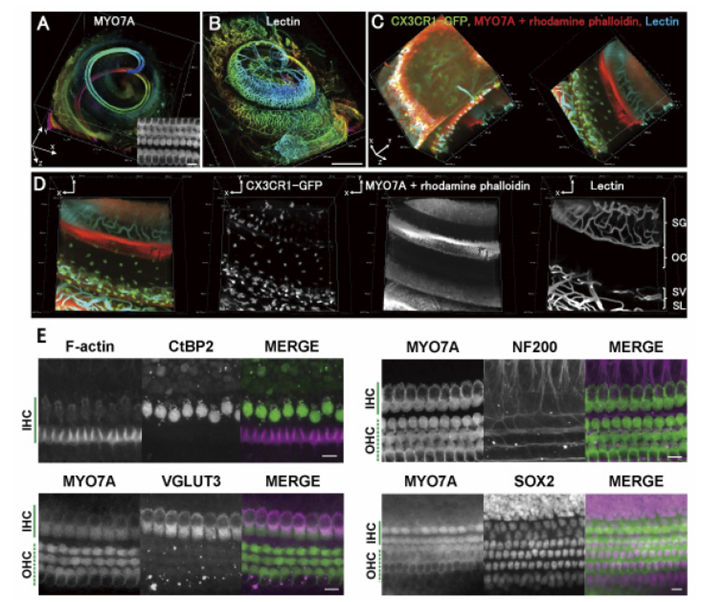
图5. 全面分析耳蜗
可观察到耳蜗内的毛细胞(A)、血管(B)。放大耳蜗管(C)则可观察到螺旋神经节(SG)、柯蒂氏器(OC)、血管纹(SV)、螺旋韧带(SL)内的血管、组织巨噬细胞(CX3CR1-GFP)。另外,可对带状突触(CtBP2)、神经丝(NF200)、谷氨酸转运蛋白(VGLUT3),支持细胞核(SOX2)等进行免疫染色。
使用显微镜:Nikon A1MP使用镜头:N25X-APO-MP(NA1.10, WD2.00), VC 20x(NA0.75, WD1.00)
◆参考文献
1)Fujimoto, C. et al. : Cell Death Dis., 8(5), e2780 (2017).
2)Mizushima, Y. et al. :Biochem. Biophys. Res. Commun., 493 (2), 894 (2017).
3)Urata, S.et al. :eLife, 8, e40946 (2019).
4)Urata, S. et al. : Bio-protoc., 9 (16), e3342 (2019).
5)Ertürk, A. et al. : Nat. Protoc., 7 (11), 1983 (2012).
6)Renier, N. et al. : Cell, 159 (4), 896 (2014).
7)Chung, K. et al. : Nature, 497, 332 (2013).
8)Susaki, EA. et al. : Cell, 157 (3), 726 (2014).
